Serwis aparatury w Katedrze Mikrobiologii na Wydziale Lekarskim UJ CM
Serwisowane urządzenie CHEF Cooling Module wchodzi w skład wyposażenia Zakładu Molekularnej Mikrobiologii Medycznej Katedry Mikrobiologii UJ CM (Ryc. 1). CHEF Cooling Module (moduł chłodzący bufor) stanowi jeden z kluczowych podzespołów unikalnego zestawu przeznaczonego do analizy restrykcyjnej chromosomalnego DNA połączonej z elektroforezą pulsową określaną skrótem REA-PFGE (ang. Restriction Endonuclease Analysis-Pulsed-Field Gel Electrophoresis). Dodatkowo w skład całej aparatury wchodzą pozostałe elementy, tj.: pompa cyrkulacyjna, komora elektroforetyczna, programowany zasilacz oraz system do wylewania żeli (Ryc. 2).


Ryc. 1. Zestaw przeznaczony do analizy restrykcyjnej chromosomalnego DNA Ryc. 2. Serwisowane urządzenie CHEF Cooling Module (moduł chłodzący bufor)
CHEF Cooling Module jest wykorzystywany w Zakładzie Molekularnej Mikrobiologii Medycznej Katedry Mikrobiologii UJ CM do realizacji szeregu projektów. Na podstawie badań prowadzonych z udziałem CHEF Cooling Module opublikowano liczne prace o zasięgu międzynarodowym. Artykuły pojawiły się m.in. w: IJERP, PJM, Folia Microbiol, Methods Mol Biol, New Microbiol, BMC Infect Dis, PLoS One, BMC Pregnancy Childbirth, BMC Microbiol.
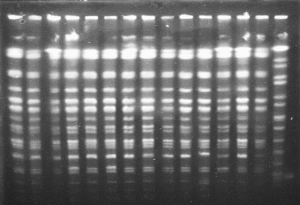
REA-PFGE jest jedną z metod biologii molekularnej uznaną za tzw. „złoty standard” w typowaniu szczepów. Charakteryzuje się wysoką powtarzalnością oraz dużą siłą dyskryminacji. REA-PFGE jest to metoda, w której oczyszczony DNA bakteryjny trawiony jest rzadko-tnącymi enzymami restrykcyjnymi (Ryc. 3). DNA wyizolowany z drobnoustrojów może być wykorzystany do badań na poziomie wewnątrzgatunkowym, dlatego badania takie pozwalają na ocenę stanu epidemiologicznego na danym terenie, ocenę rozprzestrzeniania się klonów wieloopornych.
Ryc. 3 Przykładowy wynik rozdziału otrzymanego w elektroforezie pulsacyjnej REA-PFGE, otrzymany z wykorzystaniem serwisowanego urządzenia CHEF Cooling Module
Tego rodzaju analizy mają szerokie zastosowanie w badaniach naukowych oraz w dochodzeniach epidemiologicznych stanowiąc podstawę mikrobiologicznego opracowania ogniska epidemicznego, czyli wykrycie czynnika etiologicznego, ustalenie źródła, dróg szerzenia się zakażeń, a także utrzymywanie się szczepów epidemicznych czy endemicznych. Niejednokrotnie typowanie jest wykorzystywane do identyfikacji drobnoustrojów chorobotwórczych, w sytuacji gdy cechy chorobotwórczości lub lekooporności prezentowane są przez określone klony zaliczane do danego gatunku (Ryc. 4).

Ryc. 4. Przykładowy dendrogram utworzony na podstawie żelu z metody REA-PFGE prezentujący podobieństwo genetyczne szczepów Klebsiella pneumoniae wytwarzających β-laktamazy o rozszerzonym spektrum substratowym ESβL (ang. Extended Spectrum β-Lactamases), po trawieniu enzymem restrykcyjnym SpeI, wykreślony z użyciem programu Gel Compar® II6.5 (Applied Maths). Stosując metodę REA-PFGE dla 29 badanych izolatów uzyskano 9 wzorów genetycznych (pulsotypów) oznaczonych kolejno literami (A-I), przy czym 11 (40%) szczepów zaliczono do klonu A, 6 (21%) izolatów zakwalifikowano do klonu B, 5 (17%) do klonu C, 3 (10%) izolaty do klonu D. Po 2 (7%) izolaty reprezentowały dwie mniejsze linie klonalne oznaczone literami E i F. Dla trzech szczepów (10%) uzyskano unikatowe wzory restrykcyjne oznaczone jako G, H oraz I.
Naprawa aparatury dokonana ze środków finansowych Priorytetowego Obszaru Badawczego qLIFE w ramach programu strategicznego Inicjatywa Doskonałości w Uniwersytecie Jagiellońskim.



 FACEBOOK
FACEBOOK